Biologie computationnelle : quelques questions techniques et éthiques


Bernard Ourghanlian a rejoint Microsoft France en 1999 en tant que CTO et depuis 2001 exerce également la fonction de CSO. A ce titre, il travaille sur la stratégie de Technologie et de Sécurité de Microsoft en France.
« […] Jugeons que le corps d’un homme vivant diffère autant de celui d’un homme mort que fait une montre, ou autre automate (c’est-à-dire autre machine qui se meut de soi-même), lorsqu’elle a en soi le principe corporel des mouvements pour lesquels elle est instituée, avec tout ce qui est requis pour son action, et la même montre ou autre machine, lorsqu’elle est rompue et que le principe de son mouvement cesse d’agir. »[1]
Dans ce texte célèbre, Descartes établit un parallèle, non pas entre l’homme et la machine, mais entre deux relations, celle du vivant au mort, d’une part, et celle de l’automate en état de marche au même automate cassé, de l’autre. Ici, Descartes ne dit pas explicitement que les hommes sont des automates, mais il établit une sorte d’analogie de référence ou de proportion : l’automate n’est rien d’autre qu’un certain type de machine dotée d’autonomie de mouvement ; de même, l’homme vivant correspond aussi à un corps doté de capacité de mouvement autonome, alors qu’il est inerte quand il est sans vie.
Peut-on donner la vie à une machine ? Peut-on reprogrammer cette vie et jusqu’à quel point ? Quelles sont les bases éthiques pour ce faire ? Telles sont les quelques questions auxquelles nous allons tenter de répondre. Avec beaucoup de modestie car nombre de questions aussi bien techniques qu’éthiques restent sans réponse.
La capacité de programmer la biologie pourrait constituer l’une des plus grandes révolutions technologiques du 21ème siècle. Cela pourrait permettre la réalisation de percées fondamentales dans les domaines de la médecine, de l’alimentation et de l’énergie, et jeter les bases d’une future bioéconomie.
C’est ce que l’on appelle la « biologie computationnelle » ou « biologie numérique » qui est une branche de la biologie qui implique le développement et l’application de méthodes d’analyse de données et théoriques, de modélisation mathématique et de techniques de simulation computationnelle à l’étude des systèmes biologiques, écologiques, comportementaux et sociaux. Le domaine est largement défini et comprend des fondements en biologie, mathématiques appliquées, statistiques, biochimie, chimie, biophysique, biologie moléculaire, génétique, génomique, informatique et évolution.
La biologie numérique est différente de l’informatique biologique, qui est un sous-domaine de l’informatique et de l’ingénierie informatique utilisant la bio-ingénierie et la biologie pour construire des ordinateurs, mais est similaire à la bio-informatique, qui est une science interdisciplinaire utilisant des ordinateurs pour stocker et traiter des données biologiques.[2]
Nous sommes désormais en route vers cette révolution. En effet, nous disposons désormais de la technologie pour lire et écrire des génomes entiers à un rythme qui augmente bien plus vite que la loi de Moore.
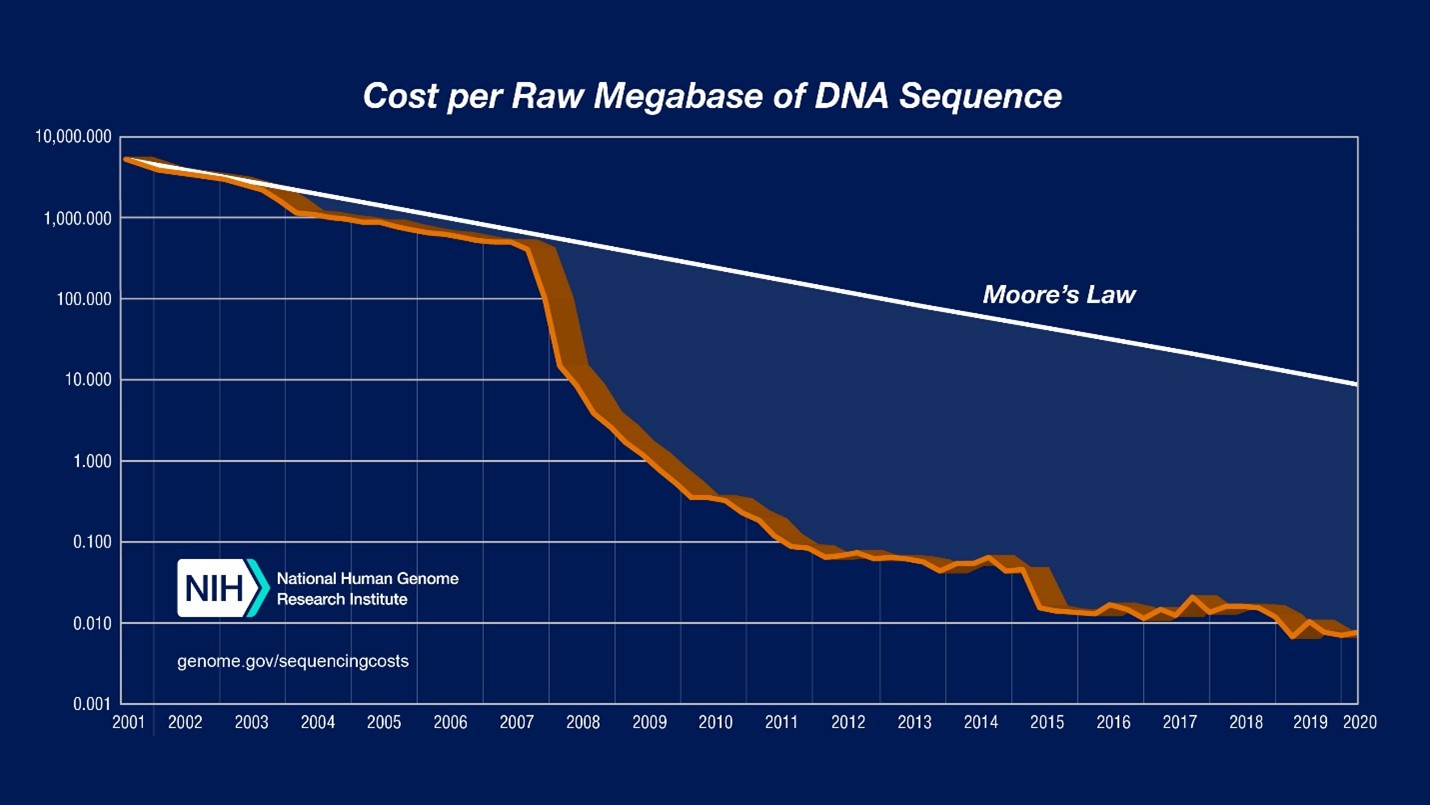
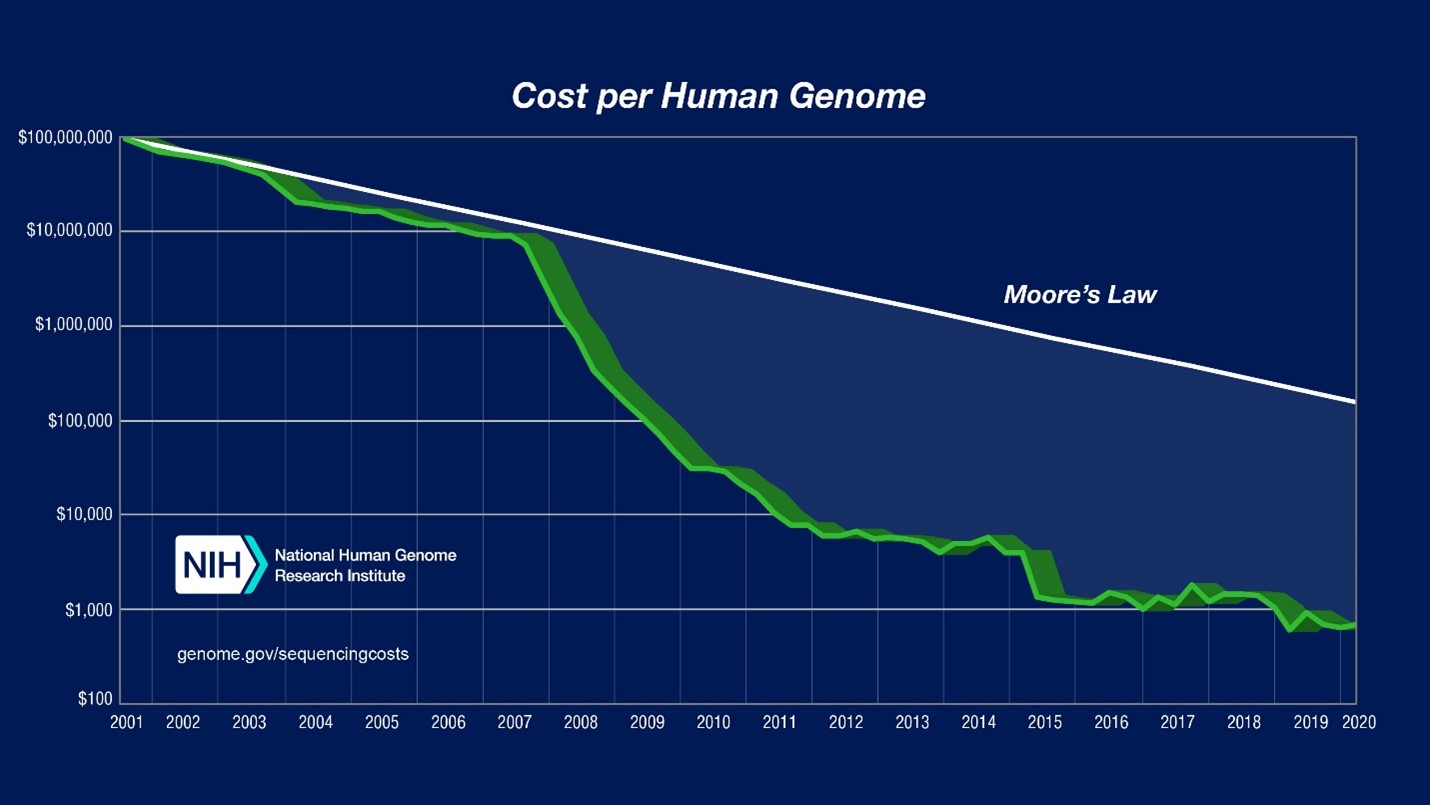
Les données de comptabilité analytique présentées ici sont articulées selon deux paramètres: (1) « Coût par mégabase de séquence d’ADN » , c’est à dire le coût de détermination d’une mégabase[3] de séquence d’ADN d’une qualité spécifiée ; (2) « Coût par génome », c’est-à-dire le coût du séquençage d’un génome de taille humaine. Pour chacune de ces données, le graphique logarithmique (sur l’axe des ordonnées, chaque nouvelle ligne équivaut à une multiplication par 10) ci-dessus présente les évolutions du coût depuis 2001.
De plus, nous pouvons désormais éditer les génomes avec une précision sans précédent, grâce à une technologie révolutionnaire appelée CRISPR / Cas9 qui a « pris d’assaut » le monde de la biologie et de la chimie ces dernières années. C’est d’ailleurs la découverte de cette technologie qui a permis à Emmanuelle Charpentier et Jennifer A. Doudna de mériter le prix Nobel de Chimie cette année[4].
Mais voyons de manière un peu plus précise de quoi il s’agit.
 Voir aussi [PODCASTS] Inside IA : L’Intelligence Artificielle à portée de tous
Voir aussi [PODCASTS] Inside IA : L’Intelligence Artificielle à portée de tous
L’approche de la biologie computationnelle
Les cellules des organismes vivants fonctionnent comme de minuscules ordinateurs, exécutant des programmes à l’échelle moléculaire pour faire de nombreuses choses étonnantes. Vous n’êtes pas convaincus… Prenez l’exemple des cellules immunitaires comme les « lymphocytes T » qui jouent un grand rôle dans la réponse immunitaire de nos organismes. Il y a même des lymphocytes « mémoire » qui sont formées lors de l’activation d’un lymphocyte T « naïf » (première infection). Ces lymphocytes T mémoires « patrouillent » dans la lymphe, les ganglions lymphatiques, le sang, la rate, etc. afin de réagir à la présence d’une infection. Et comme leur seuil d’activation est plus faible comparé aux lymphocytes T naïfs, ceci rend la réponse mémoire bien plus rapide et efficace.
Ces cellules doivent résoudre le problème de la lutte contre les maladies et les infections sans lutter contre le corps lui-même, et c’est typiquement un problème de traitement de l’information. Donc, d’une manière ou d’une autre, il doit y avoir un programme fonctionnant à l’intérieur de ces cellules, un programme qui répond aux signaux d’entrée et façonne ce que cette cellule pourrait faire, un programme d’interactions dynamiques à l’échelle moléculaire. Et ces programmes doivent fonctionner de manière distribuée dans tout notre système immunitaire, en coordonnant et en partageant les informations pour protéger tout notre corps. Il est donc possible de considérer un tel fonctionnement comme celui d’un logiciel vivant. Si nous pouvions comprendre les programmes biologiques qui fonctionnent à l’intérieur des cellules, comme ceux de notre système immunitaire, cela transformerait notre capacité à comprendre comment et pourquoi les cellules font ce qu’elles font. Et si nous comprenions vraiment ces programmes biologiques, alors nous pourrions les déboguer lorsque les choses tournent mal. Et nous pourrions réaliser des choses vraiment incroyables… En restant conscient – et nous y reviendrons – du fait que, pour rendre hommage à Bernard Stiegler, tristement disparu cet été : « tout objet technique est pharmacologique : il est à la fois poison et remède. Le pharmakon est à la fois ce qui permet de prendre soin et ce dont il faut prendre soin, au sens où il faut y faire attention : c’est une puissance curative dans la mesure et la démesure où c’est une puissance destructrice. Cet « à la fois » est ce qui caractérise la pharmacologie qui tente d’appréhender par le même geste le danger et ce qui sauve. Toute technique est originairement et irréductiblement ambivalente »[5]. Ces choses incroyables peuvent donc également se transformer en cauchemar.
Pour revenir au sujet, chaque organisme vivant est fabriqué de cellules. Et chaque cellule contient des molécules d’ADN, qui codent les instructions génétiques contrôlant le fonctionnement de ces cellules.
Vous vous demandez peut-être pourquoi Microsoft s’intéresse à la compréhension de la biologie computationnelle. En fait, c’est tout simple : l’informatique est rendue possible en encodant des uns et des zéros au sein de puces de silicium. Quelque chose de similaire se produit dans les cellules des organismes vivants, mais le « code » ici est représenté par des G, C, A et T, c’est-à-dire par les éléments constitutifs de l’ADN.[6] Et c’est à travers la confrontation de domaines scientifiques distincts que l’on arrive à faire progresser à la fois ces différents domaines ; ici, l’informatique, la biologie et les mathématiques.

Chez Microsoft, une équipe connue sous le nom de « Station B » a travaillé avec des partenaires industriels et des startups pour tirer parti de notre expertise dans la création de langages de programmation et de compilateurs, du Machine Learning et du Cloud pour développer des outils qui permettront aux scientifiques de découvrir comment s’opère le calcul au sein des systèmes vivants et de faire de la biologie numérique une technique aussi sûre et robuste que la programmation logicielle traditionnelle.
Pour traduire les conceptions de haut niveau du comportement biologique en circuits pouvant être assemblés en laboratoire, nous avons besoin de l’équivalent d’un « compilateur génétique ». Ainsi, tout comme le développement logiciel est aujourd’hui fiable et reproductible – dans une large mesure, même s’il y a toujours des bogues – nos outils permettront aux scientifiques de concevoir des circuits génétiques, de les construire en laboratoire, de tester s’ils produisent le bon comportement et d’apprendre de ces expériences.
Avec ces outils, les scientifiques peuvent envisager de concevoir des circuits génétiques pour modifier le comportement des cellules. À l’avenir, par exemple, ces technologies pourraient, par exemple, aider à traiter un patient atteint d’Alzheimer en stimulant de nouvelles cellules cérébrales, ou permettre aux plantes de résister à certains ravageurs et d’offrir ainsi des rendements plus élevés.
 Lire aussi [TECH AU CARRE] L’informatique quantique n’aura plus de secret pour vous
Lire aussi [TECH AU CARRE] L’informatique quantique n’aura plus de secret pour vous
Les difficultés à résoudre
Aujourd’hui, nous ne comprenons que partiellement comment programmer la biologie. Ceci peut être attribué à trois limitations principales :
Premièrement, nous ne disposons pas d’une compréhension fondamentale de la façon dont les systèmes biologiques « calculent ». En termes simples, il est difficile de programmer un système dont nous ne comprenons pas suffisamment le fonctionnement. En effet, les systèmes biologiques sont très complexes : ils sont massivement parallèles, distribués, probabilistes et à bien des égards beaucoup plus proches des ordinateurs analogiques que des ordinateurs numériques. « Les ordinateurs numériques traitent des nombres entiers, des séquences binaires, de la logique déterministe, des algorithmes, ainsi que du temps conçu comme une séquence d’événements discrets. Les ordinateurs analogiques traitent de nombres réels, d’une logique non déterministe et de fonctions continues, et du temps tel qu’il existe dans le monde réel, comme un continuum. En informatique analogique, la complexité réside dans la topologie et non dans le code. »[7]
Plus généralement, nous avons encore un long chemin à parcourir pour rétro-concevoir ces systèmes, qui n’ont pas été conçus par nous mais par des milliards d’années d’évolution.
Deuxièmement, nous manquons de méthodes systématiques pour programmer le calcul en biologie. Par conséquent aujourd’hui, la programmation de la biologie se fait principalement par une séries d’essais et d’erreurs.
Enfin, nous manquons de protocoles robustes d’automatisation de laboratoire. De telle sorte que la plupart des expériences sont effectuées manuellement, prennent du temps et sont sujettes aux erreurs. Ainsi, des études récentes ont montré que la plupart des expériences biologiques ne sont pas reproductibles, avec des taux d’impossibilité de reproduction pouvant atteindre jusqu’à 89% dans certains cas[8]
À bien des égards, l’état actuel de la biologie computationnelle ressemble beaucoup aux débuts de la programmation sur ordinateur, avant l’existence de langages de haut niveau, de compilateurs et d’une grande partie de la théorie fondamentale de l’informatique que nous tenons désormais pour acquise.
Au sein de Microsoft Research, nous travaillons à relever ces défis fondamentaux à travers l’étude de la biologie numérique :
Tout d’abord, nous faisons progresser notre compréhension du calcul biologique en identifiant les structures de données moléculaires, les algorithmes biologiques et les principes fondamentaux du calcul en biologie ; en effet, nous disposons de tels principes pour le calcul au sein d’un ordinateur, mais pas encore pour le calcul biologique.
Ensuite, nous développons des méthodes de programmation du calcul en biologie, et notamment des langages de programmation, des compilateurs, des méthodes de vérification et de débogage – autant de choses que nous tenons actuellement pour acquises en informatique – ainsi que des caractérisations détaillées des composants biologiques, analogues aux fiches techniques utilisées dans le monde numérique.
Enfin, nous développons des protocoles d’automatisation de laboratoire et avons mis en place un laboratoire humide[9] au sein de Microsoft Research pour les tester. Nous nous sommes également associés à Synthace[10], une startup d’automatisation des processus de laboratoire basée sur le Cloud Azure, qui a développé le langage Antha pour l’encodage numérique des expériences biologiques. Ce langage fonctionne sur une large gamme de robots de laboratoire, y compris les nôtres.
L’intérêt de la création d’une plateforme
Nous avons également pris la décision d’intégrer l’ensemble de nos méthodes au sein d’une plate-forme complète pour la biologie computationnelle, avec pour objectif à long terme de faire passer cette biologie numérique d’un processus d’essais-erreurs que nous avons évoqués plus haut en un processus beaucoup plus systématique et prévisible. Les principales caractéristiques de cette plateforme sont les suivantes :
Essentiellement, pour programmer la biologie, nous devons comprendre :
- Comment programmer des molécules d’ADN ;
- Comment programmer des dispositifs génétiques insérés dans les cellules pour en modifier le comportement cellulaire ;
- Et comment procéder à l’ingénierie inverse de types de cellules spécifiques, comme les cellules souches, afin que nous puissions les reprogrammer pour des applications ciblées, telles que la médecine régénérative.
Nous avons donc développé des DSL (Domain Specific Language) dans ces trois domaines et publié nos résultats dans des revues scientifiques de premier plan[11].
Fondamentalement, différents programmes biologiques nécessitent différents niveaux d’abstraction, de sorte que notre plate-forme prend en charge une large gamme d’abstractions, ainsi que des compilateurs pour réaliser la traduction entre les abstractions et des méthodes de vérification et d’analyse pour chacune des abstractions. Par exemple, une chaîne de Markov à temps continu[12] peut être utilisée pour déterminer comment le bruit aléatoire affecte le fonctionnement du système, en utilisant la simulation stochastique ou des méthodes de vérification de modèle. De même, les autres abstractions ont leurs propres méthodes correspondantes[13].
Nous intégrons ces abstractions au sein de méthodes d’automatisation utilisées au sein de notre propre laboratoire humide, ainsi que des techniques avancées d’analyse de données s’appuyant sur une infrastructure de robotique de laboratoire et reposant sur les capacités de stockage et de calcul du Cloud Azure.
On voit donc se dessiner en filigrane l’architecture d’une plate-forme[14] visant à améliorer toutes les phases du workflow Conception-Construction-Test-Apprentissage qui est généralement utilisé pour la programmation de systèmes biologiques :
- La phase de conception intégrera des langages de programmation biologique qui opèrent aux niveaux moléculaire, génétique et réseau. Ces langages peuvent à leur tour être compilés en une hiérarchie d’abstractions biologiques, chacune avec leurs méthodes d’analyse associées, où différentes abstractions pouvant être sélectionnées en fonction de la question biologique abordée.
- La phase de construction intégrera des compilateurs qui traduisent des programmes de haut niveau en code ADN, ainsi qu’un codage numérique des expériences biologiques à effectuer.
- La phase de test exécutera des expériences biologiques à l’aide de robots de laboratoire en collaboration avec Synthace, en utilisant leur logiciel primé Antha, une puissante plate-forme logicielle basée sur l’Internet des Objets construite dans le Cloud Azure qui donne aux biologistes un contrôle sophistiqué sur le matériel de laboratoire.
- La phase d’apprentissage intégrera une gamme de méthodes pour extraire des connaissances biologiques à partir de données expérimentales, et notamment l’inférence bayésienne, le raisonnement symbolique et les méthodes d’apprentissage en profondeur, fonctionnant à grande échelle sur Azure.
Ces phases seront intégrées à une base de connaissances biologiques qui stocke des modèles de calcul représentant la compréhension actuelle des systèmes biologiques considérés. Au fur et à mesure que de nouvelles expériences seront effectuées, la base de connaissances sera mise à jour via un apprentissage automatisé.
Programmation de dispositifs génétiques
La programmation des dispositifs génétiques est réalisée en insérant de l’ADN dans les cellules pour en modifier le comportement. Cette approche sous-tend la majeure partie de la bioéconomie actuelle.
Nous pouvons considérer l’ADN comme le code machine de la cellule, qui définit le code pour les protéines qui déterminent finalement le comportement de la cellule.
Ce code peut être divisé en instructions génétiques, représentées par des identifiants, où chaque identifiant représente une séquence d’ADN distincte. Ceci est analogue à l’utilisation des noms d’instructions dans le langage machine de l’ordinateur, pour représenter une séquence binaire correspondante.
L’ADN dispose de la notion de « promoteur », ou « séquence promotrice », qui est une région de l’ADN située à proximité d’un gène et est indispensable à la transcription de l’ADN en ARN. Le promoteur est la zone de l’ADN sur laquelle se fixe initialement l’ARN polymérase, avant de démarrer la synthèse de l’ARN. Les séquences promotrices sont en général situées en amont du démarrage de la transcription[15]. De même, la notion de terminateur ou « terminateur de transcription » est une séquence du génome qui marque la fin de la transcription d’un gène ou en ARN messager par l’ARN polymérase[16]. Ceci permet d’utiliser les instructions du promoteur et du terminateur pour indiquer où la cellule doit commencer et arrêter la lecture de l’ADN. Cela se réalise à l’aide d’une molécule spéciale appelée polymérase, pour produire de l’ARN.
Ces différents éléments peuvent souvent nous aider, étant donné une courte séquence d’ADN, à déterminer comment elle sera exécutée dans une cellule. On voit que ces éléments ne sont vraiment pas loin du fonctionnement d’une machine de Turing et que ceux-ci peuvent nous aider à envisager de programmer des dispositifs génétiques.
Cependant, lors de la programmation de la biologie, nous voulons généralement accomplir le processus inverse : définir d’abord le comportement souhaité puis déterminer la séquence d’ADN nécessaire pour implémenter ce comportement.
Pour cela, Microsoft Research a développé un langage de programmation de cellules. Le programmeur écrit une description de haut niveau du comportement souhaité de la cellule (d’où l’importance des DSL dont nous parlions plus haut qui ont été définis à cette fin) et un compilateur génère automatiquement le code ADN nécessaire pour implémenter ce comportement.
Nous avons même utilisé notre langage et nos méthodes de caractérisation pour programmer, pour la première fois, la communication entre cellules sur deux canaux indépendants, une sorte de TCP/IP pour les cellules. En effet, la communication cellulaire est fondamentale à la biologie, de l’organisation des colonies de cellules microbiennes au développement de l’organisme, en passant par le fonctionnement du système immunitaire qui est essentiellement distribué.
Mais en termes de capacité à programmer la communication cellulaire, jusqu’à présent, l’état de l’art était de programmer des cellules pour communiquer sur un seul canal dans une seule direction. Les tentatives de mise en œuvre de la communication sur plusieurs canaux étaient limitées par les interférences entre les molécules de la cellule. Ce type d’interférence constitue, en effet, l’un des défis fondamentaux de la programmation de la biologie, car les molécules d’une cellule existent souvent au sein d’une « soupe chimique » et peuvent interférer au hasard les unes avec les autres.
Microsoft Research a réalisé une percée remarquable dans ce domaine en permettant la mise en œuvre, pour la première fois, d’une communication cellulaire programmée sur deux canaux indépendants.[17] Cela nécessitait des modèles informatiques détaillés de dispositifs génétiques, paramétrés par des données expérimentales. Nous avons construit des appareils récepteurs et émetteurs indépendants et les avons utilisés pour démontrer un contrôle précis de la communication cellulaire dans le temps et dans l’espace.
Un tel contrôle précis de la communication cellulaire a de nombreuses applications potentielles, de la programmation de cellules microbiennes pour optimiser la production de médicaments et de carburants, à la programmation de cellules dans un tissu pour former des organes ou des matériaux complexes qui s’auto-assemblent et se réparent, à la programmation de nos cellules immunitaires pour cibler plus efficacement des maladies comme le cancer.
Et après ?
Microsoft a annoncé la création de « Station B » à Cambridge en Angleterre au mois de mars 2019 afin d’aller de l’avant dans le domaine de la biologie computationnelle. Le nom « Station B » est directement inspiré de « Station Q », qui a lancé les efforts de Microsoft dans le domaine de l’informatique quantique.
Ainsi, de la même façon que « Station Q » vise à construire un ordinateur quantique, « Station B » vise à construire une plate-forme pour la programmation de la biologie. Bien que nous ayons déjà fait des progrès significatifs dans ce sens, il reste, en effet, encore beaucoup à faire.
« Station B » fait partie de cet effort plus large, avec un accent sur le développement d’une plate-forme intégrée qui permet à des partenaires sélectionnés d’améliorer la productivité au sein de leurs propres organisations, conformément à la mission de Microsoft. Le projet Station B s’appuie sur plus d’une décennie de recherche chez Microsoft portant sur la compréhension et la programmation du traitement de l’information au sein des systèmes biologiques, en collaboration avec plusieurs universités de premier plan. Pour en apprendre plus sur ce programme, il est possible d’écouter le podcast et de parcourir le site https://innovation.microsoft.com/en-us/biological-computation.
La capacité de programmer la biologie pourrait permettre des percées fondamentales au sein d’un large éventail d’industries, depuis la médecine à l’agriculture en passant par l’alimentation, la construction, les textiles, les matériaux et les produits chimiques. Cela pourrait également aider à jeter les bases d’une future bioéconomie basée sur une technologie durable.
Ainsi, pour nous lancer dans un peu de prospective, que se passerait-il si nous pouvions nous programmer pour détecter, éradiquer ou même prévenir les maladies mieux que nous ne le pouvons actuellement ou même remplacer les tissus endommagés ? Cela signifierait vivre plus longtemps en meilleure santé. Ou que se passerait-il si nous pouvions programmer les cultures pour résister aux pathogènes fongiques ou même pour porter des fruits plus souvent ? Cela nous permettrait de nourrir une planète affamée. Ou si nous pouvions trouver comment exploiter les effets quantiques pour capturer l’énergie du soleil de manière vraiment efficace, plus efficace que nos panneaux solaires actuels ? Cela pourrait devenir l’énergie verte ultime… Il est difficile de ne pas être enthousiasmé par de telles potentialités.
Malgré cet énorme potentiel et ces belles promesses, la programmation de la biologie se fait encore aujourd’hui en grande partie sur la base d’essais et d’erreurs. Pour relever ce défi, le domaine de la biologie computationnelle travaille collectivement depuis près de deux décennies pour développer de nouvelles méthodes et technologies de programmation de la biologie.
Par ailleurs, et bien qu’il y ait beaucoup d’idées fausses sur la biologie en général et sur la biologie computationnelle en particulier, celle-ci présente cependant de véritables défis en matière d’éthique dont on ne peut pas faire l’économie.
 Lire aussi « La technologie doit servir la vision du monde que nous souhaitons pour demain »
Lire aussi « La technologie doit servir la vision du monde que nous souhaitons pour demain »
Et l’éthique dans tout cela ?
On a dit que la biologie synthétique représentait le deuxième saut quantique en matière d’éthique en biologie… Le premier s’est produit lorsque les scientifiques ont commencé à modifier génétiquement la vie en recombinant l’ADN de différentes espèces il y a 30 ans. En biologie synthétique, les scientifiques peuvent maintenant introduire dans les blocs de construction des organismes des éléments qui n’y auraient jamais été sans cela grâce une combinaison de matériaux existants. La création du premier organisme – une cellule bactérienne – contrôlée par un génome synthétisé chimiquement a été rapportée par le J. Graig Venter Institute en 2010. Des chercheurs du Venter Institute ont synthétisé le génome de la bactérie Mycoplasma mycoides en construisant des enregistrements informatiques de sa structure puis en construisant un réplique du génome à partir d’éléments inanimés. Une fois le génome terminé, il a été transplanté dans une cellule de Mycoplasma capricolum dont l’ADN a été retiré. La bactérie produite, Synthia, était capable de se répliquer encore et encore ; par conséquent, elle était par définition viable et vivante[18]. Les gros titres annonçaient alors : « Les biologistes synthétiques jouent à Dieu »[19].
Les questions philosophiques et éthiques qui ont été soulevées au cours de l’histoire de la biologie moléculaire et de la génétique sont encore prégnantes à l’ère de la biologie computationnelle. Trois de ces préoccupations semblent émerger. La première est que la création de nouvelles formes de vie est ambivalente : soit intrinsèquement mauvaise, soit pouvant engendrer des conséquences néfastes (cf. le commentaire plus haut sur la notion de pharmakon). La seconde est qu’aujourd’hui, le travail ou la réglementation sur le terrain sont conduits soit par une peur injustifiée, soit par un optimisme irraisonné. Et la troisième est que l’ingénierie de la vie, comme toutes les technologies émergentes, a besoin de l’approbation la plus large des citoyens pour pouvoir se dérouler sans heurts. Il suffit, pour s’en convaincre, de voir comment le débat sur les OGM a prospéré en France et en Europe.
Les éthiciens favorables au progrès la science s’appuient ce que l’on pourrait appeler le « principe de l’espoir ». Celui-ci procède de la conviction que le progrès technologique est essentiel pour le bien-être futur de l’humanité et que les obstacles fondés sur les peurs et les angoisses causent plus de souffrance que ne le feraient même les inventions les plus dangereuses. C’est la poursuite de la vision des Modernes qui avaient érigé en principe fondateur le culte de l’avenir et la foi inébranlable dans le progrès. J’en veux pour preuve les déclarations de Condorcet : « La nature n’a mis aucun terme à nos espérances » ou de Saint-Simon[20] : « L’âge d’or du genre humain n’est point derrière nous, il est au-devant, il est dans la perfection de l’ordre social », ou encore de Victor Hugo[21] : « L’épanouissement du genre humain de siècle en siècle, l’homme montant des ténèbres à l’idéal, la transfiguration paradisiaque de l’enfer terrestre, l’éclosion lente et suprême de la liberté, droit pour cette vie, responsabilité pour l’autre […]. » Ces deux mamelles de la modernité – le culte de l’avenir et la foi en un progrès qui n’aurait pas de fin – ont nourri en leur sein des générations successives de scientifiques et d’informaticiens dont la mienne…
La prescription qui en découle est que, étant donné que la biologie synthétique peut conduire à des développements bénéfiques, elle devrait être autorisée à se poursuivre, sous contrôle éthique, mais autrement sans entrave, à moins qu’il ne puisse être démontré de manière fiable qu’elle n’est pas sûre.
La logique sous-jacente des deux principes est similaire et peut être attribuée à « l’argument du pari » introduit par Blaise Pascal[22] pour prouver si Dieu existe ou non : nous avons des raisons rationnelles de croire que tel est le cas, et de nous comporter en conséquence. Si Dieu existe et que nous le concédons, notre récompense en sera la béatitude éternelle. Si Dieu existe et que nous le nions, notre châtiment sera un tourment éternel. Les enjeux sont, en d’autres termes, très importants. Mais pas dans le cas contraire… Même si Dieu n’existait pas, nous ne gagnerions pas grand-chose à professer un univers impie : seulement quelques plaisirs transitoires que nous pourrions éprouver en ne nous soumettant pas aux règles bibliques. En tant que joueurs rationnels, nous devons comprendre que, quelles que soient les probabilités, les profits et les pertes infinis l’emportent sur les finis. L’infini divisé par un nombre fini est toujours l’infini. Dans l’incertitude, nous devrions, selon Pascal, choisir l’engagement avec des coûts finis et des gains infinis, ou au contraire, ne pas choisir un engagement avec des gains finis (permission de commettre quelques péchés) et des coûts potentiellement infinis (souffrance éternelle dans l’au-delà).
Les comparaisons entre le pari de Pascal et les principes de précaution et d’espoir révèlent où se situe le poids réel des arguments. Pour Pascal, la promesse ou la menace de l’au-delà était suffisamment réelle pour justifier sa conclusion. Des personnes plus laïques pourraient objecter en notant que les « quelques plaisirs éphémères » auxquels il fait allusion constituent la vie humaine, la seule existence que nous connaissons, qui est trop précieuse pour être jetée dans l’espoir ou la crainte d’une possibilité logique d’un autre genre. d’existence. De même, la menace de catastrophe causée par le génie génétique ou la biologie synthétique est suffisamment réelle et importante pour que les partisans du principe de précaution bloquent le progrès scientifique et technologique, tandis que l’espoir de bonheur pousse les champions du principe d’espoir à plaider pour des restrictions minimales. sur les avancées émergentes. Pour mémoire, les prémices modernes du « principe de précaution » viennent d’Allemagne, dans le courant des années 1970 : Vorsorgeprinzip (« principe de prévoyance » ou « principe de souci »). Il a été popularisé par le philosophe Hans Jonas dans « Le Principe responsabilité » (1979). Pour Jonas, la puissance technologique moderne pose de nouveaux problèmes éthiques. Les hommes doivent ainsi exiger le risque zéro de conduire à la destruction des conditions d’une vie authentiquement humaine sur Terre. Jonas entend s’opposer à ceux qui considèrent la Terre et ses habitants comme un objet avec lequel toutes les expérimentations sont possibles, sur le plan juridique comme sur le plan moral.[23]
Je ne me lancerai pas ici dans un débat philosophique sur les avantages et les inconvénients du « principe de précaution » qui pourrait nous entrainer trop loin.
Idéalement, si nous nous engageons à respecter les valeurs d’autonomie, de démocratie et d’égalité, nous devrions tous être bien informés de ce qui se passe dans le domaine des technologies émergentes, nous devrions tous avoir notre mot à dire sur la façon dont elles sont développées, et nous devrions tous bénéficier de leurs avancées. Les comités d’éthique, les commissions et les groupes de travail sur ce type de sujet ne sont pas toujours suffisamment équipés pour atteindre ces objectifs alors que les technologies sous-jacentes sont très complexes et font souvent appel à des domaines scientifiques multiples ; par conséquent, il faut trouver autre chose. Mais il n’est pas encore clair de savoir quelle pourrait être cette chose…
Dans une logique d’une éthique conséquentialiste[24] qui, dans le contexte de l’économie de l’innovation, tend à dominer les situations d’évaluation des développements technologiques, on peut se dire que les risques de la biologie computationnelle dépassent les gains… Ou bien le contraire…
Lorsqu’Hannah Arendt nous dit que « c’est dans le vide de pensée que s’inscrit le mal »[25], elle ne veut pas dire que les gens mauvais sont des idiots « par nature », mais tout simplement que ce sont des personnes qui n’ont pas exercé leurs facultés de jugement moral. Dans le cas qui nous intéresse ici à propos de la biologie computationnelle, il est essentiel que chacun de nous, en tant que citoyen, exerce ces facultés de jugement moral. Et pour cela, en préalable, une information didactique auprès de chacun de nos concitoyens, mêlant à la fois l’informatique, la biologie et les mathématiques, est absolument nécessaire.
« Si vous trouvez que l’éducation coûte cher, essayez l’ignorance. »[26] La biologie computationnelle ne pourra pas faire l’impasse sur l’éducation si elle souhaite réaliser les promesses qu’elle envisage. En permettant la libre évaluation des options et le libre choix parmi les possibles…
[1] Les Passions de l’âme, René Descartes, Première Partie, Article VI
[2] Source : https://fr.wikipedia.org/wiki/Biologie_num%C3%A9rique
[3] La mégabase (Mb) est une unité de mesure en biologie moléculaire représentant une longueur d’un million de paires de bases d’ADN ou d’ARN.
[4] https://www.nobelprize.org/prizes/chemistry/2020/press-release/
[5] http://arsindustrialis.org/pharmakon
[6] L’ADN est constitué de deux chaînes (ou brins) enroulées l’une autour de l’autre en une double hélice de 2 nm (10-9 m) de diamètre. Chaque chaîne est composée d’une succession de nucléotides qui sont un assemblage de trois molécules : un groupement phosphate, un sucre (désoxyribose) et une base azotée. Les bases azotées sont au nombre de quatre : adénine (notée A), thymine (T), guanine (G), cytosine ©. La complémentarité de ces bases, deux à deux, permet l’association des deux brins d’ADN : l’adénine est toujours appariée à une thymine et la guanine à une cytosine. Source : CNRS
[7] https://www.edge.org/conversation/george_dyson-childhoods-end
[8] https://journals.plos.org/plosbiology/article?id=10.1371/journal.pbio.1002165
[9] Un laboratoire humide est un type de laboratoire dans lequel un large éventail d’expériences sont effectuées, par exemple, la caractérisation d’enzymes en biologie, le titrage en chimie, la diffraction de la lumière en physique, etc. Ces expériences peuvent parfois impliquer de traiter des substances dangereuses. En raison de la nature de ces expériences, la disposition appropriée des équipements de sécurité est d’une grande importance.
[10] https://www.synthace.com/customers/case-studies/detail/construct-assembly-with-microsoft-research/
[11] Domain-specific programming languages for computational nucleic acid systems
[12] https://fr.wikipedia.org/wiki/Processus_de_Markov_%C3%A0_temps_continu
[13] https://www.microsoft.com/en-us/research/project/stationb/
[14] Plus d’informations sur cette plateforme en https://www.microsoft.com/en-us/research/project/stationb/
[15] Source : https://fr.wikipedia.org/wiki/Promoteur_(biologie)
[16] Source : https://fr.wikipedia.org/wiki/Terminateur_(g%C3%A9n%C3%A9tique)
[17] Voir https://www.embopress.org/doi/full/10.15252/msb.20156590 et https://www.microsoft.com/en-us/research/publication/dna-based-communication-in-populations-of-synthetic-protocells/
[18] https://en.wikipedia.org/wiki/Mycoplasma_laboratorium
[19] https://www.telegraph.co.uk/news/science/7745868/Scientist-Craig-Venter-creates-life-for-first-time-in-laboratory-sparking-debate-about-playing-god.html
[20] Claude-Henri de Saint Simon, 1814.
[21] Victor Hugo, 1859.
[22] https://fr.wikipedia.org/wiki/Pari_de_Pascal
[23] Source : https://fr.wikipedia.org/wiki/Principe_de_pr%C3%A9caution
[24] Le conséquentialisme désigne une démarche qui détermine le caractère bon ou mauvais d’une intention ou d’une action en regard de leurs conséquences, c’est-à-dire, d’abord, de leurs effets observables et évaluables d’un point de vue donné, ce dernier pouvant varier.
[25] Hannah Arendt « Les origines du totalitarisme », Tome 3 : Le système totalitaire – 1951
[26] Les versions apocryphes attribuent cette citation à Abraham Lincoln mais il semble que la paternité de celle-ci revienne à Robert Orben (cf. https://dicocitations.lemonde.fr/citations/citation-58292.php).

 Temps de lecture : 21 minutes
Temps de lecture : 21 minutes